Demo data
The package includes data from the CG-Tch site for
testing and tutorial purposes, as well an internal site list derived
from the FluxnetEO publication (including small corrections as
presented).
The internal site list can be called using:
library(FluxnetEO)
head(fluxnet_sites)
#> site_code latitude longitude
#> 1 AR-SLu −33.4648 −66.4598
#> 2 AT-Neu 47.1167 11.3175
#> 3 AU-Ade −13.0769 131.1178
#> 4 AU-Cum −33.6152 150.7236
#> 5 AU-DaS −14.1593 131.3881
#> 6 AU-Emr −23.8587 148.4746Larger demo data is stored in the package data directory (see below).
Reading FluxnetEO netcdf summary data
The main functionality of this small package is to provide an easy routine to access FluxnetEO data in R for further synthesis. The below code shows you how to read in the cutout data from a given path. Note that this routine executed on a directory containing for example the MODIS data will summarize this data in one big tidy data frame (~3 GB in memory, 600 MB as compressed RDS file).
# list all cutout summary files
files <- list.files(
system.file(package = "FluxnetEO", "extdata"),
utils::glob2rx("*_cutout.nc"),
full.names = TRUE,
recursive = TRUE
)
# loop over all files and bind
# them in one data frame
data <- lapply(files, function(file){
feo_read_nc(
file = file
)
})
data <- do.call("rbind", data)
# print the data
print(head(data))
#> EVI EVI_N EVI_NSTD EVI_gapfilltype NDVI NDVI_N NDVI_NSTD NDVI_gapfilltype
#> 1 NaN 0 NaN 999 NaN 0 NaN 999
#> 2 NaN 0 NaN 999 NaN 0 NaN 999
#> 3 NaN 0 NaN 999 NaN 0 NaN 999
#> 4 NaN 0 NaN 999 NaN 0 NaN 999
#> 5 NaN 0 NaN 999 NaN 0 NaN 999
#> 6 NaN 0 NaN 999 NaN 0 NaN 999
#> NDWI_N NDWI_NSTD NIRv NIRv_N NIRv_NSTD NIRv_gapfilltype kNDVI kNDVI_N
#> 1 0 NaN NaN 0 NaN 999 NaN 0
#> 2 0 NaN NaN 0 NaN 999 NaN 0
#> 3 0 NaN NaN 0 NaN 999 NaN 0
#> 4 0 NaN NaN 0 NaN 999 NaN 0
#> 5 0 NaN NaN 0 NaN 999 NaN 0
#> 6 0 NaN NaN 0 NaN 999 NaN 0
#> kNDVI_NSTD kNDVI_gapfilltype sWDRVI sWDRVI_N sWDRVI_NSTD sWDRVI_gapfilltype
#> 1 NaN 999 NaN 0 NaN 999
#> 2 NaN 999 NaN 0 NaN 999
#> 3 NaN 999 NaN 0 NaN 999
#> 4 NaN 999 NaN 0 NaN 999
#> 5 NaN 999 NaN 0 NaN 999
#> 6 NaN 999 NaN 0 NaN 999
#> NDWI_SWIR1 NDWI_SWIR1_N NDWI_SWIR1_NSTD NDWI_SWIR1_gapfilltype NDWI_SWIR2
#> 1 NaN 0 NaN 999 NaN
#> 2 NaN 0 NaN 999 NaN
#> 3 NaN 0 NaN 999 NaN
#> 4 NaN 0 NaN 999 NaN
#> 5 NaN 0 NaN 999 NaN
#> 6 NaN 0 NaN 999 NaN
#> NDWI_SWIR2_N NDWI_SWIR2_NSTD NDWI_SWIR2_gapfilltype NDWI_SWIR3 NDWI_SWIR3_N
#> 1 0 NaN 999 NaN 0
#> 2 0 NaN 999 NaN 0
#> 3 0 NaN 999 NaN 0
#> 4 0 NaN 999 NaN 0
#> 5 0 NaN 999 NaN 0
#> 6 0 NaN 999 NaN 0
#> NDWI_SWIR3_NSTD NDWI_SWIR3_gapfilltype RED RED_N RED_NSTD RED_gapfilltype
#> 1 NaN 999 NaN 0 NaN 999
#> 2 NaN 999 NaN 0 NaN 999
#> 3 NaN 999 NaN 0 NaN 999
#> 4 NaN 999 NaN 0 NaN 999
#> 5 NaN 999 NaN 0 NaN 999
#> 6 NaN 999 NaN 0 NaN 999
#> NIR_N NIR NIR_NSTD NIR_gapfilltype BLUE BLUE_N BLUE_NSTD BLUE_gapfilltype
#> 1 0 NaN NaN 999 NaN 0 NaN 999
#> 2 0 NaN NaN 999 NaN 0 NaN 999
#> 3 0 NaN NaN 999 NaN 0 NaN 999
#> 4 0 NaN NaN 999 NaN 0 NaN 999
#> 5 0 NaN NaN 999 NaN 0 NaN 999
#> 6 0 NaN NaN 999 NaN 0 NaN 999
#> GREEN GREEN_N GREEN_NSTD GREEN_gapfilltype SWIR1 SWIR1_N SWIR1_NSTD
#> 1 NaN 0 NaN 999 NaN 0 NaN
#> 2 NaN 0 NaN 999 NaN 0 NaN
#> 3 NaN 0 NaN 999 NaN 0 NaN
#> 4 NaN 0 NaN 999 NaN 0 NaN
#> 5 NaN 0 NaN 999 NaN 0 NaN
#> 6 NaN 0 NaN 999 NaN 0 NaN
#> SWIR1_gapfilltype SWIR2 SWIR2_N SWIR2_NSTD SWIR2_gapfilltype SWIR3 SWIR3_N
#> 1 999 NaN 0 NaN 999 NaN 0
#> 2 999 NaN 0 NaN 999 NaN 0
#> 3 999 NaN 0 NaN 999 NaN 0
#> 4 999 NaN 0 NaN 999 NaN 0
#> 5 999 NaN 0 NaN 999 NaN 0
#> 6 999 NaN 0 NaN 999 NaN 0
#> SWIR3_NSTD SWIR3_gapfilltype LST_TERRA_Day LST_N_TERRA_Day LST_NSTD_TERRA_Day
#> 1 NaN 999 307.3877 0 NaN
#> 2 NaN 999 307.3877 0 NaN
#> 3 NaN 999 307.3877 0 NaN
#> 4 NaN 999 307.3877 0 NaN
#> 5 NaN 999 307.3877 0 NaN
#> 6 NaN 999 307.3877 0 NaN
#> LST_gapfilltype_TERRA_Day LST_AQUA_Day LST_N_AQUA_Day LST_NSTD_AQUA_Day
#> 1 4 311.5907 0 NaN
#> 2 4 311.5907 0 NaN
#> 3 4 311.5907 0 NaN
#> 4 4 311.5907 0 NaN
#> 5 4 311.5907 0 NaN
#> 6 4 311.5907 0 NaN
#> LST_gapfilltype_AQUA_Day LST_TERRA_Night LST_N_TERRA_Night
#> 1 4 295.1425 0
#> 2 4 295.1425 0
#> 3 4 295.1425 0
#> 4 4 295.1425 0
#> 5 4 295.1425 0
#> 6 4 295.1425 0
#> LST_NSTD_TERRA_Night LST_gapfilltype_TERRA_Night LST_AQUA_Night
#> 1 NaN 4 288.2467
#> 2 NaN 4 288.2467
#> 3 NaN 4 288.2467
#> 4 NaN 4 288.2467
#> 5 NaN 4 288.2467
#> 6 NaN 4 288.2467
#> LST_N_AQUA_Night LST_NSTD_AQUA_Night LST_gapfilltype_AQUA_Night
#> 1 0 NaN 4
#> 2 0 NaN 4
#> 3 0 NaN 4
#> 4 0 NaN 4
#> 5 0 NaN 4
#> 6 0 NaN 4
#> LST_TERRA_Day_VZA0 LST_N_TERRA_Day_VZA0 LST_NSTD_TERRA_Day_VZA0
#> 1 309.5584 0 NaN
#> 2 309.5584 0 NaN
#> 3 309.5584 0 NaN
#> 4 309.5584 0 NaN
#> 5 309.5584 0 NaN
#> 6 309.5584 0 NaN
#> LST_gapfilltype_TERRA_Day_VZA0 LST_AQUA_Day_VZA0 LST_N_AQUA_Day_VZA0
#> 1 4 311.1001 0
#> 2 4 311.1001 0
#> 3 4 311.1001 0
#> 4 4 311.1001 0
#> 5 4 311.1001 0
#> 6 4 311.1001 0
#> LST_NSTD_AQUA_Day_VZA0 LST_gapfilltype_AQUA_Day_VZA0 LST_TERRA_Night_VZA0
#> 1 NaN 4 295.5699
#> 2 NaN 4 295.5699
#> 3 NaN 4 295.5699
#> 4 NaN 4 295.5699
#> 5 NaN 4 295.5699
#> 6 NaN 4 295.5699
#> LST_N_TERRA_Night_VZA0 LST_NSTD_TERRA_Night_VZA0
#> 1 0 NaN
#> 2 0 NaN
#> 3 0 NaN
#> 4 0 NaN
#> 5 0 NaN
#> 6 0 NaN
#> LST_gapfilltype_TERRA_Night_VZA0 LST_AQUA_Night_VZA0 LST_N_AQUA_Night_VZA0
#> 1 4 289.202 0
#> 2 4 289.202 0
#> 3 4 289.202 0
#> 4 4 289.202 0
#> 5 4 289.202 0
#> 6 4 289.202 0
#> LST_NSTD_AQUA_Night_VZA0 LST_gapfilltype_AQUA_Night_VZA0 LST_TERRA_Day_VZA40
#> 1 NaN 4 307.2047
#> 2 NaN 4 307.2047
#> 3 NaN 4 307.2047
#> 4 NaN 4 307.2047
#> 5 NaN 4 307.2047
#> 6 NaN 4 307.2047
#> LST_N_TERRA_Day_VZA40 LST_NSTD_TERRA_Day_VZA40
#> 1 0 NaN
#> 2 0 NaN
#> 3 0 NaN
#> 4 0 NaN
#> 5 0 NaN
#> 6 0 NaN
#> LST_gapfilltype_TERRA_Day_VZA40 LST_AQUA_Day_VZA40 LST_N_AQUA_Day_VZA40
#> 1 4 310.3047 0
#> 2 4 310.3047 0
#> 3 4 310.3047 0
#> 4 4 310.3047 0
#> 5 4 310.3047 0
#> 6 4 310.3047 0
#> LST_NSTD_AQUA_Day_VZA40 LST_gapfilltype_AQUA_Day_VZA40 LST_TERRA_Night_VZA40
#> 1 NaN 4 295.0825
#> 2 NaN 4 295.0825
#> 3 NaN 4 295.0825
#> 4 NaN 4 295.0825
#> 5 NaN 4 295.0825
#> 6 NaN 4 295.0825
#> LST_N_TERRA_Night_VZA40 LST_NSTD_TERRA_Night_VZA40
#> 1 0 NaN
#> 2 0 NaN
#> 3 0 NaN
#> 4 0 NaN
#> 5 0 NaN
#> 6 0 NaN
#> LST_gapfilltype_TERRA_Night_VZA40 LST_AQUA_Night_VZA40 LST_N_AQUA_Night_VZA40
#> 1 4 288.7238 0
#> 2 4 288.7238 0
#> 3 4 288.7238 0
#> 4 4 288.7238 0
#> 5 4 288.7238 0
#> 6 4 288.7238 0
#> LST_NSTD_AQUA_Night_VZA40 LST_gapfilltype_AQUA_Night_VZA40 date site
#> 1 NaN 4 2000-01-01 CG-Tch
#> 2 NaN 4 2000-01-02 CG-Tch
#> 3 NaN 4 2000-01-03 CG-Tch
#> 4 NaN 4 2000-01-04 CG-Tch
#> 5 NaN 4 2000-01-05 CG-Tch
#> 6 NaN 4 2000-01-06 CG-TchPlot data
# convert date string to date object
# for nice plotting
data <- data %>%
mutate(
date = as.Date(date)
)
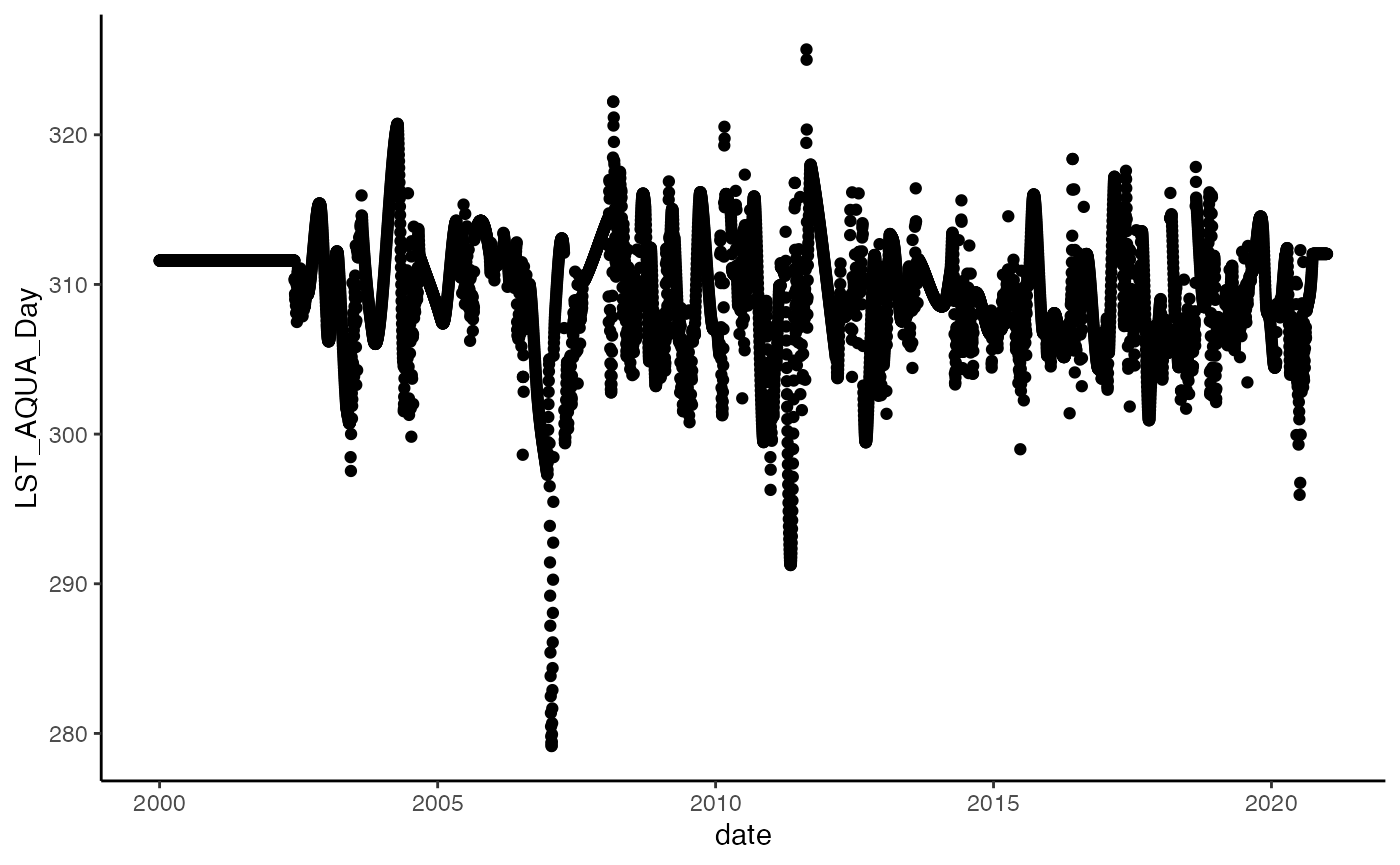
# quick ggplot graph of the MODIS Aqua daytime
# land surface temperature (LST)
ggplot(data) +
geom_point(
aes(
date,
LST_AQUA_Day
)
) +
theme_classic()